Bakterielle Infektionen
Wir arbeiten an der schnellen Identifizierung bakterieller Erreger samt ihres Antibiotikaresistenz-Profils
Bakterien-Spezies und -Antibiotikaresistenzen
Unsere Kartusche
amp.flow ist mit einer Amplifikationskammer, einem Sensorbereich zum Anbringen eines Objektträgers, der ein Microarray mit Fängersonden enthält, sowie mit zahlreichen Reservoirs für die Lagerung von Reagenzien ausgestattet.
Amplifikation, Markierung und Hybridisierung können auf der Kartusche stattfinden und mit einem automatisierten Protokoll ablaufen.
Pumpen, Temperieren und Spot-Detektion sind in dem
universellen Kartuschenlesegerät UCAR
kombiniert, wodurch manuelle Schritte minimiert werden.
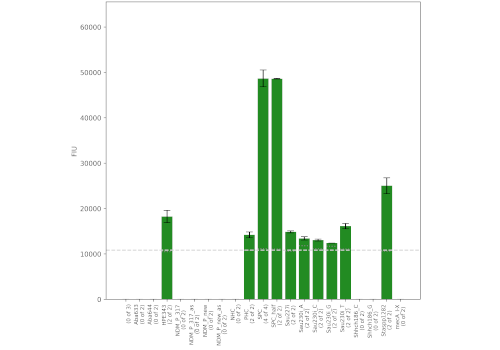
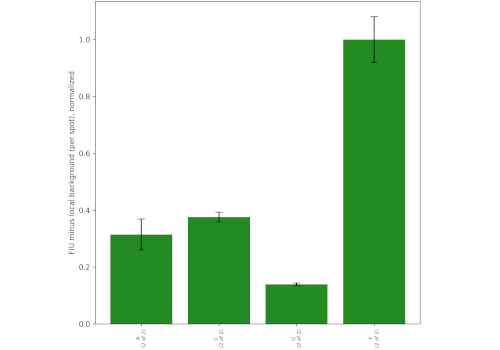
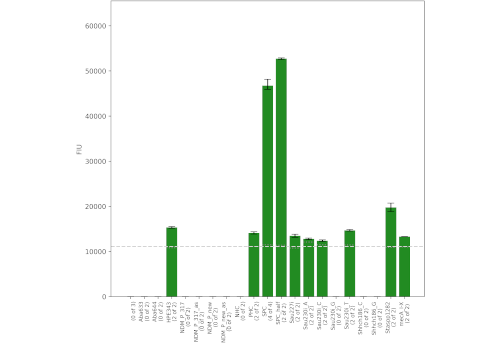
Die folgenden Beispiele zeigen den erfolgreichen Nachweis von PCR-Produkten von
- A. baumannii mit NDM-Resistenz (Neu-Delhi-Metallo-beta-Lactamase) sowie
- S. aureus ohne Methicillin-Resistenz (MSSA) und
- S. aureus mit Methicillin-Resistenz (MRSA)
- S. hominis
mit Methicillin-Resistenz
unter Nutzung desselben Microarray-Layouts und Assays.
Die Kartuschen wurden von unserem universellen Kartuschenlesegerät UCAR kontrolliert und ausgelesen. Das Microarray wurde automatisch verarbeitet, um die Spot-Signale zu quantifizieren und anschließend die Spezies und Resistenzen zu bestimmen. Für S. aureus wurde ein Einzelnukleotid-Polymorphismus (SNP) nachgewiesen, um S. aureus von anderen Staphylococcus-Arten zu unterscheiden. S. hominis wird ebenfalls selektiv mit einem zusätzlichen SNP-Sonden-Set detektiert.
Beispielhafte Ergebnisse:
Art: Acinetobacter baumannii
Resistenz: NDM


Art: Staphylococcus aureus
Resistenz: -

Art: Staphylococcus aureus
Resistenz: mecA (MRSA)


Art: Staphylococcus hominis
Resistenz: mecA






